Sitzung 5
Wiederholung
Das Wichtigste vom letzten Mal
- Wir haben uns verschiedene Funktionen angesehen, die in der Statistik benutzt werden
Das Wichtigste vom letzten Mal
- Wir haben gelernt, Häufigkeiten zählen zu lassen
Das Wichtigste vom letzten Mal
- Wir haben uns die Struktur von Datenblättern angesehen
## carat cut color clarity depth table price x y z
## 1 0.23 Ideal E SI2 61.5 55 326 3.95 3.98 2.43
## 2 0.21 Premium E SI1 59.8 61 326 3.89 3.84 2.31
## 3 0.23 Good E VS1 56.9 65 327 4.05 4.07 2.31## 'data.frame': 53940 obs. of 10 variables:
## $ carat : num 0.23 0.21 0.23 0.29 0.31 0.24 0.24 0.26 0.22 0.23 ...
## $ cut : Ord.factor w/ 5 levels "Fair"<"Good"<..: 5 4 2 4 2 3 3 3 1 3 ...
## $ color : Ord.factor w/ 7 levels "D"<"E"<"F"<"G"<..: 2 2 2 6 7 7 6 5 2 5 ...
## $ clarity: Ord.factor w/ 8 levels "I1"<"SI2"<"SI1"<..: 2 3 5 4 2 6 7 3 4 5 ...
## $ depth : num 61.5 59.8 56.9 62.4 63.3 62.8 62.3 61.9 65.1 59.4 ...
## $ table : num 55 61 65 58 58 57 57 55 61 61 ...
## $ price : int 326 326 327 334 335 336 336 337 337 338 ...
## $ x : num 3.95 3.89 4.05 4.2 4.34 3.94 3.95 4.07 3.87 4 ...
## $ y : num 3.98 3.84 4.07 4.23 4.35 3.96 3.98 4.11 3.78 4.05 ...
## $ z : num 2.43 2.31 2.31 2.63 2.75 2.48 2.47 2.53 2.49 2.39 ...Das Wichtigste vom letzten Mal
## carat cut color clarity depth
## Min. :0.2000 Fair : 1610 D: 6775 SI1 :13065 Min. :43.00
## 1st Qu.:0.4000 Good : 4906 E: 9797 VS2 :12258 1st Qu.:61.00
## Median :0.7000 Very Good:12082 F: 9542 SI2 : 9194 Median :61.80
## Mean :0.7979 Premium :13791 G:11292 VS1 : 8171 Mean :61.75
## 3rd Qu.:1.0400 Ideal :21551 H: 8304 VVS2 : 5066 3rd Qu.:62.50
## Max. :5.0100 I: 5422 VVS1 : 3655 Max. :79.00
## J: 2808 (Other): 2531
## table price x y
## Min. :43.00 Min. : 326 Min. : 0.000 Min. : 0.000
## 1st Qu.:56.00 1st Qu.: 950 1st Qu.: 4.710 1st Qu.: 4.720
## Median :57.00 Median : 2401 Median : 5.700 Median : 5.710
## Mean :57.46 Mean : 3933 Mean : 5.731 Mean : 5.735
## 3rd Qu.:59.00 3rd Qu.: 5324 3rd Qu.: 6.540 3rd Qu.: 6.540
## Max. :95.00 Max. :18823 Max. :10.740 Max. :58.900
##
## z
## Min. : 0.000
## 1st Qu.: 2.910
## Median : 3.530
## Mean : 3.539
## 3rd Qu.: 4.040
## Max. :31.800
## Tabellen miteinander verbinden
Verbinden von zwei Tabellen
- Tabellen untereinander zusammenfügen:
- Tabellen nebeneinander zusammenfügen:
Übung
- Lest die folgenden Tabellen in R ein
- Briefe.csv
- Briefe2.csv
- Tagebuecher.csv
- Tagebuecher2.csv
- Verbindet alle Tabellen sinnvoll miteinander
Lösung
- Lest die Tabellen in R ein
d1 <- read.csv("docs/data/tut5/Briefe.csv", sep = ";", dec = ",")
d2 <- read.csv("docs/data/tut5/Briefe2.csv", sep = ";", dec = ",")
d3 <- read.csv("docs/data/tut5/Tagebuecher.csv", sep = ";", dec = ",")
d4 <- read.csv("docs/data/tut5/Tagebuecher2.csv", sep = ";", dec = ",")
head(d1, 3)## Filename Segment WC WPS Sixltr Pronoun I We
## 1 m-Rollet_Alexander-BR-1844_1906.txt 1 500 19.23 32.4 4.0 0.0 0.4
## 2 m-Rollet_Alexander-BR-1844_1906.txt 2 500 18.52 32.8 7.6 3.6 0.6
## 3 m-Rollet_Alexander-BR-1844_1906.txt 3 500 41.67 28.8 9.2 0.0 5.6
## Self You
## 1 0.4 2.2
## 2 4.2 1.6
## 3 5.6 0.8## Other Preps Affect Posemo Negemo Past Present Future
## 1 3.6 9.4 3.2 2.4 0.8 1.2 1.6 0.2
## 2 3.2 7.2 3.6 3.2 0.2 2.2 2.2 0.2
## 3 3.2 7.2 2.0 2.0 0.0 2.4 2.8 1.4Lösung
- Verbindet alle Tabellen sinnvoll miteinander
dneu1 <- cbind(d1, d2) # schreibt die Tabellen nebeneinander
dneu2 <- cbind(d3, d4) # schreibt die Tabellen nebeneinander
dneuges <- rbind(dneu1, dneu2) # schreibt die Tabellen untereinander
head(dneuges)## Filename Segment WC WPS Sixltr Pronoun I We
## 1 m-Rollet_Alexander-BR-1844_1906.txt 1 500 19.23 32.4 4.0 0.0 0.4
## 2 m-Rollet_Alexander-BR-1844_1906.txt 2 500 18.52 32.8 7.6 3.6 0.6
## 3 m-Rollet_Alexander-BR-1844_1906.txt 3 500 41.67 28.8 9.2 0.0 5.6
## 4 m-Rollet_Alexander-BR-1844_1906.txt 4 500 31.25 36.2 5.8 0.0 1.2
## 5 m-Rollet_Alexander-BR-1844_1906.txt 5 500 23.81 34.8 5.0 0.0 0.6
## 6 m-Rollet_Alexander-BR-1844_1906.txt 6 500 25.00 36.0 8.4 4.8 0.6
## Self You Other Preps Affect Posemo Negemo Past Present Future
## 1 0.4 2.2 3.6 9.4 3.2 2.4 0.8 1.2 1.6 0.2
## 2 4.2 1.6 3.2 7.2 3.6 3.2 0.2 2.2 2.2 0.2
## 3 5.6 0.8 3.2 7.2 2.0 2.0 0.0 2.4 2.8 1.4
## 4 1.2 1.8 4.4 7.2 3.8 3.4 0.4 2.4 3.4 2.4
## 5 0.6 2.2 3.4 6.6 3.4 3.4 0.0 1.6 2.2 0.6
## 6 5.4 1.6 2.6 11.4 4.6 3.8 0.8 1.8 3.6 0.2Fehlende Werte
NAs
Fehlende Werte führen dazu, dass Berechnungen ins Leere laufen
## [1] NA## [1] NAZwar ist dies bei intern erstellten Daten kein Problem, aber was ist, wenn Werte in einem Datenset fehlen wie hier:
## id geo subject variant experiment item condition judgement
## 1 m-1987-EMusik bla 1 1 9 4 a 6
## 2 m-1987-EMusik bla 1 1 8 2 b 7
## 3 m-1987-EMusik bla 1 1 7 10 b 7
## 4 m-1987-EMusik bla 1 1 9 3 d 1
## 5 m-1987-EMusik bla 1 1 1 8 d NA
## 6 m-1987-EMusik bla 1 1 8 9 a 6## [1] NANAs finden
anyNA gibt an, ob fehlende Werte enthalten sind:
## [1] TRUE## [1] TRUEis.na geht das ganze Objekt durch, durchsucht es nach NAs und gibt deren Präsenz über Wahrheitswerte an:
## [1] FALSE TRUE FALSE FALSEDa Tabellen in der Regel sehr viele Werte enthalten, macht es hier Sinn, sich das Ergebnis der Suche als Tabelle ausgeben zu lassen:
##
## FALSE TRUE
## 3836 4alle Zeilen mit NAs
## id geo subject variant experiment item condition judgement
## 5 m-1987-EMusik bla 1 1 1 8 d NA
## 205 m-1983-EnglSport bla 3 1 7 12 d NA
## 2431 m-86-psych ??? 31 6 7 12 c NA
## 3284 m-1987-geschengl ??? 42 7 8 22 d NAProzent NA
Zeigt für jede Spalte an, wie viel Prozent der Daten NAs sind
## id geo subject variant experiment item condition
## 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000 0.0000000
## judgement
## 0.1041667NAs überspringen
Möglichkeit 1:
## [1] 4.64025Möglichkeit 2: na.rm ist ein logisches Argument, das in vielen Funktionen zusätzlich angegeben werden kann, um fehlende Werte bei der Berechnung auszusparen
## [1] 4.64025## [1] 2.435801Faktoren
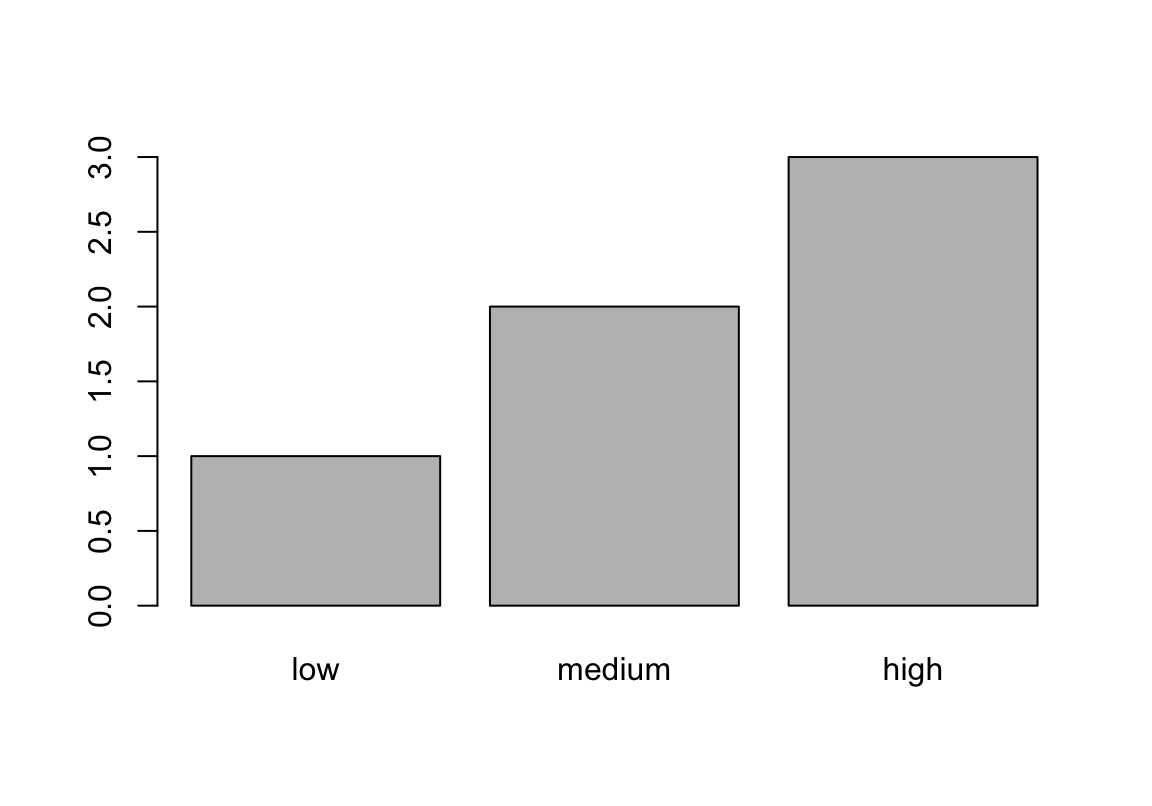
Vektoren mit Faktorstufen
Vor allem wichtig in der statistischen Berechnung und bei Plots, weil Faktoren in R der Datentyp von kategorialen und ordinalen Variablen sind (für letzteres eigentlich ordered = TRUE, kann aber regelmäßig weggelassen werden)
## NULL## [1] "high" "low" "medium"## [1] "low" "medium" "high"Level bleiben erhalten
Selbst wenn alle Instanzen eines Levels wegfallen, bleibt das Level im Faktor enthalten:
## [1] low medium medium
## Levels: low medium highDies kann mithilfe von einer erneuten Anwendung von factor behoben werden. Alternativ kann auch die droplevels-Funktion verwendet werden.
## [1] low medium medium
## Levels: low mediumKonvertierungen
weitere wichtige Konvertierungen:
- as.numeric()
- as.logical()
- as.data.frame()## Factor w/ 2 levels "low","medium": 1 2 2## chr [1:3] "low" "medium" "medium"## Factor w/ 2 levels "low","medium": 1 2 2summary()
calories <- factor(c("high", "high", "low", "medium", "high", "medium"),
levels = c("low", "medium", "high"))
summary(calories) # funktioniert wie table()## low medium high
## 1 2 3## Length Class Mode
## 6 character characterFakoren in eingelesenen Daten
R konvertiert strings, also alphabetische Zeichenketten, automatisch zu Faktoren, wenn sie in eingelesenen Daten vorkommen. Um dies zu verhindern, verwendet man stringsAsFactors = F
Übungen II
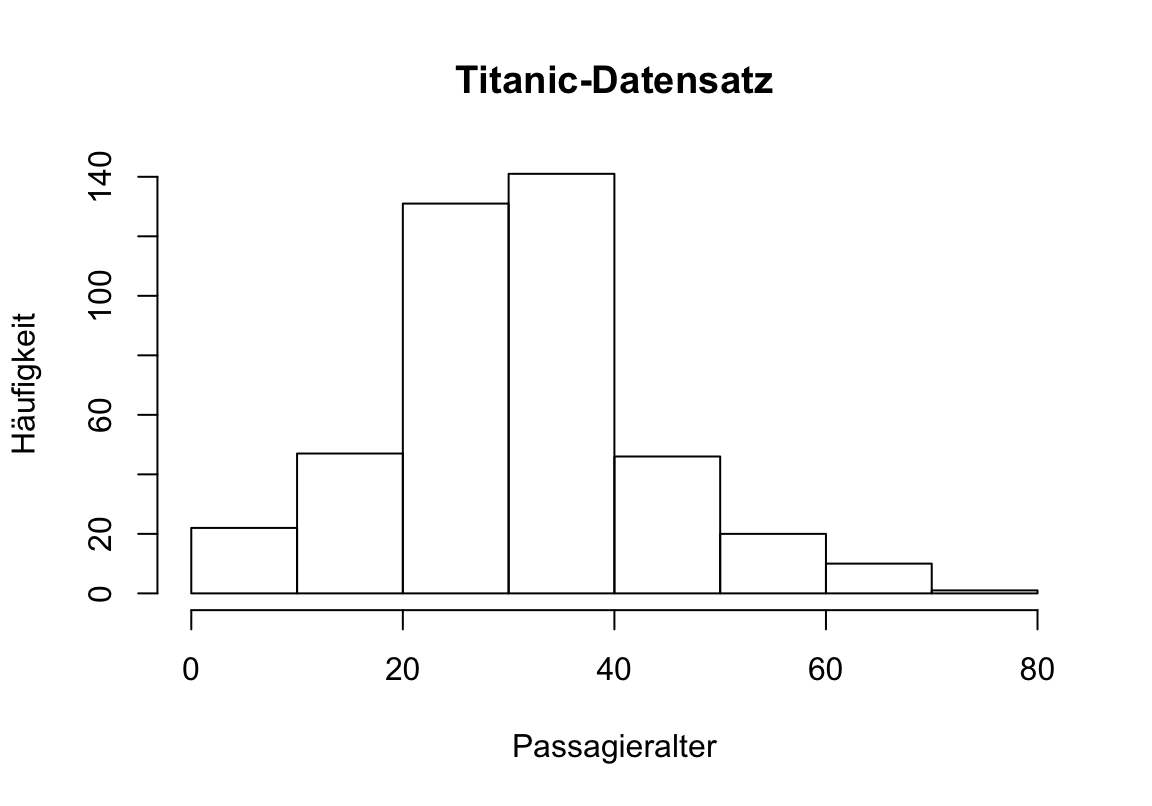
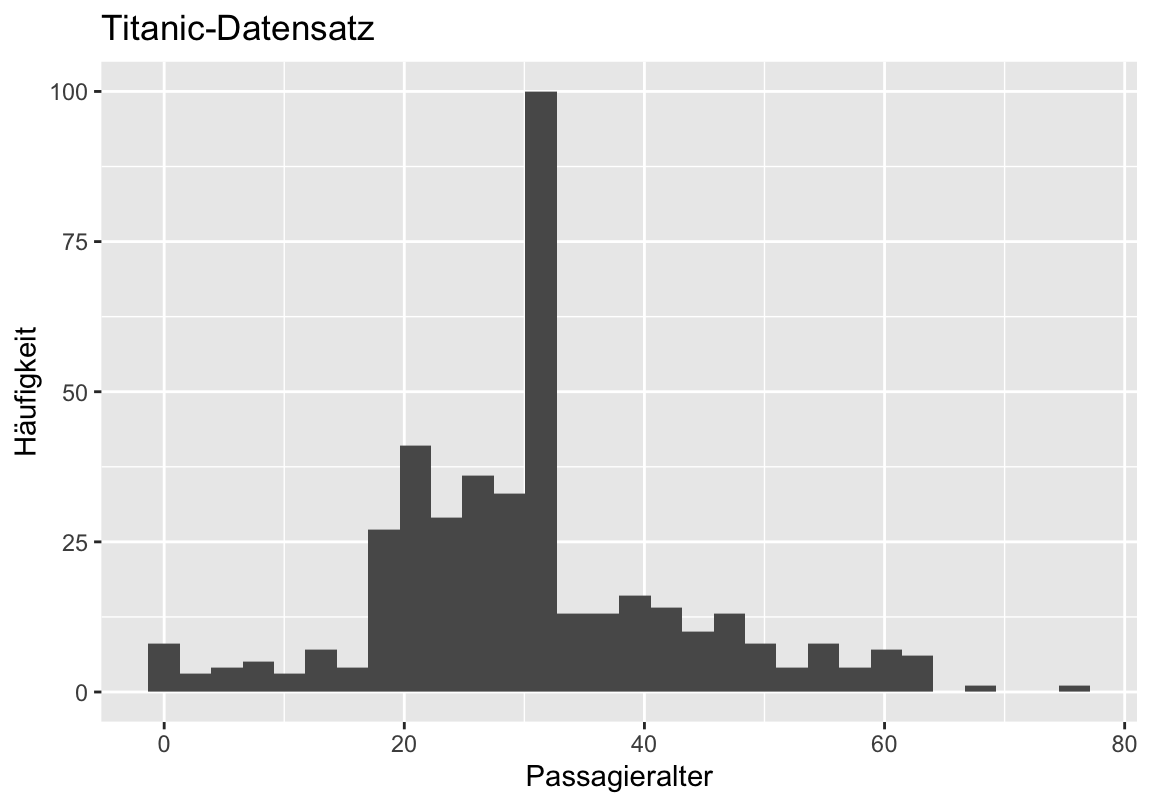
- Lese die Datei “titanic.csv” in R ein und verschaffe dir einen Überblick über die Daten
- Erstelle ein neues Objekt, das keine NAs enthält
- Ersetze im Originalobjekt alle NAs durch den Mittelwert der jeweiligen Spalte
Lösungen II
- Lese die Datei “titanic.csv” in R ein und verschaffe dir einen Überblick über die Daten
## PassengerId Pclass Name Sex Age SibSp Parch
## 1 892 3 Kelly, Mr. James male 34.5 0 0
## 2 893 3 Wilkes, Mrs. James (Ellen Needs) female 47.0 1 0
## 3 894 2 Myles, Mr. Thomas Francis male 62.0 0 0
## Ticket Fare Cabin Embarked
## 1 330911 7.8292 Q
## 2 363272 7.0000 S
## 3 240276 9.6875 Q## 'data.frame': 418 obs. of 11 variables:
## $ PassengerId: int 892 893 894 895 896 897 898 899 900 901 ...
## $ Pclass : int 3 3 2 3 3 3 3 2 3 3 ...
## $ Name : Factor w/ 418 levels "Abbott, Master. Eugene Joseph",..: 210 409 273 414 182 370 85 58 5 104 ...
## $ Sex : Factor w/ 2 levels "female","male": 2 1 2 2 1 2 1 2 1 2 ...
## $ Age : num 34.5 47 62 27 22 14 30 26 18 21 ...
## $ SibSp : int 0 1 0 0 1 0 0 1 0 2 ...
## $ Parch : int 0 0 0 0 1 0 0 1 0 0 ...
## $ Ticket : Factor w/ 363 levels "110469","110489",..: 153 222 74 148 139 262 159 85 101 270 ...
## $ Fare : num 7.83 7 9.69 8.66 12.29 ...
## $ Cabin : Factor w/ 77 levels "","A11","A18",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ Embarked : Factor w/ 3 levels "C","Q","S": 2 3 2 3 3 3 2 3 1 3 ...Lösungen II
- Erstelle ein neues Objekt, das keine NAs enthält
##
## FALSE TRUE
## 4511 87##
## FALSE TRUE
## 332 86## [1] 418 11## [1] 331 11Lösungen II
- Ersetze im Originalobjekt alle NAs durch den Mittelwert der jeweiligen Spalte
## PassengerId Pclass Name Sex
## 11 902 3 Ilieff, Mr. Ylio male
## 23 914 1 Flegenheim, Mrs. Alfred (Antoinette) female
## 30 921 3 Samaan, Mr. Elias male
## 34 925 3 Johnston, Mrs. Andrew G (Elizabeth Lily" Watson)" female
## 37 928 3 Roth, Miss. Sarah A female
## 153 1044 3 Storey, Mr. Thomas male
## Age SibSp Parch Ticket Fare Cabin Embarked
## 11 NA 0 0 349220 7.8958 S
## 23 NA 0 0 PC 17598 31.6833 S
## 30 NA 2 0 2662 21.6792 C
## 34 NA 1 2 W./C. 6607 23.4500 S
## 37 NA 0 0 342712 8.0500 S
## 153 60.5 0 0 3701 NA Stitanic$Age[is.na(titanic$Age)] <- mean(titanic$Age, na.rm = T)
titanic$Fare[is.na(titanic$Fare)] <- mean(titanic$Fare, na.rm = T)
anyNA(titanic)## [1] FALSE