Sitzung 9
Experimentanalyse
Der Plan
Heute will ich mit euch so tun, als würden wir gemeinsam ein komplettes Experiment analysieren. Das dient einerseits der Vorbereitung auf die Prüfungsleistung, wo ihr genau das machen müsst, andererseits ist es als kleine Wiederholungssitzung gedacht. Ihr könnt also überprüfen, welche Bereiche aus dem R-Handwerkszeug, das hier bereits Stoff war, schon bei euch sitzen und welche ihr euch vielleicht nochmal angucken solltet.
Genauer werden wir Folgendes machen:
- kurze Einführung in das Experimentdesign
- Rohdaten einlesen
- Daten für die Analyse aufbereiten
- überflüssige Spalten entfernen
- unabhängige Variablen codieren
- Spalten zu Faktoren umwandeln
- Deskriptive Statistik
- (Visualisierung)
- (ein kleines bisschen) Inferenzstatistik
Einführung ins Experiment
Yuqiu Chen, Mailin Antomo und ich haben ein Experiment zu folgender Fragestellung durchgeführt:
Warum gibt es einen Unterschied zwischen den beiden Satzpaaren:
- Did somebody steal a car?
- # It was the duck \([_{PSP}\) who stole a car\(]\).-Cleft
- Did somebody steal a car?
- I know \([_{PSP}\) that the duck stole a car\(]\).
Scheinbar verhalten sich nicht alle präsuppositionsauslösenden Ausdücke gleich in Bezug auf at-issueness. Können wir diesen intuitiven Unterschied empirisch festigen und auf die soft-hard-Dichotomie von Präsuppositionsauslösern zurückführen?
Einführung ins Experiment
Faktoren (unabhängige Variablen):
- : Hard Trigger (-Clefts und Adverbien) vs Soft Trigger (faktive Verben) vs Appositiver Relativsatz
- : At-Issue vs Non-At-Issue
- : Erwachsene vs Kinder
Methode (abhängige Variable):
- Akzeptabilitätsrating (1 bis 5) des b-Satzes in Bezug auf die Frage in a
Daten einlesen und aufbereiten
Daten einlesen und Überblick verschaffen
## V1 V2 V3 V4 V5 V6 V7
## 1 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796407.625 data
## 2 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796437.5646 data
## 3 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796471.0036 data
## 4 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796501.9807 data
## 5 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796538.352 data
## 6 id 3536fed0235c7b34a33ddf06fb91b390 ip ::1 time(s) 1558796576.8658 data
## V8 V9 V10 V11 V12 V13 V14 V15 V16 V17 age months gender
## 1 schaffen4_at 5 NA 0 0 NA NA 0 mailin NA 5 1 w
## 2 schaffen1_non 5 NA 0 0 NA NA 0 mailin NA 5 1 w
## 3 cleft2_non 5 NA 0 0 NA NA 0 mailin NA 5 1 w
## 4 appRel3_non 5 NA 0 0 NA NA 0 mailin NA 5 1 w
## 5 appRel9_non 4 NA 0 0 NA NA 0 mailin NA 5 1 w
## 6 gewinnen2_non 4 NA 0 0 NA NA 0 mailin NA 5 1 wÜberblick II
## V1 V2 V3 V4
## id:1181 0up71acsusa12ruvaf3llod4h6 : 30 ip:1181 ::1:1181
## 28af308e70043f33346261c3fe3a79b8: 30
## 2oocalgtfmpphi3ro9s68fu990 : 30
## 3536fed0235c7b34a33ddf06fb91b390: 30
## 3gkj6ts9s53vtfhk8v1r4d07r1 : 30
## 3jtpb1nfmpi5qb2fe95qktc8j5 : 30
## (Other) :1001
## V5 V6 V7 V8
## time(s):1181 15.431.601.928.574: 1 data:1181 auch3_at : 39
## 15.431.602.323.662: 1 appRel10_at : 26
## 15.431.603.044.837: 1 appRel3_non : 26
## 15.431.603.431.064: 1 appRel9_non : 26
## 15.431.603.785.808: 1 gewinnen2_non: 26
## 15.431.604.161.861: 1 schaffen2_at : 26
## (Other) :1175 (Other) :1012
## V9 V10 V11 V12 V13
## Min. :1.000 Mode:logical Min. :0 Min. :0 Mode:logical
## 1st Qu.:2.000 NA's:1181 1st Qu.:0 1st Qu.:0 NA's:1181
## Median :4.000 Median :0 Median :0
## Mean :3.466 Mean :0 Mean :0
## 3rd Qu.:5.000 3rd Qu.:0 3rd Qu.:0
## Max. :5.000 Max. :0 Max. :0
##
## V14 V15 V16 V17 age
## Mode:logical Min. :0 Imke :180 Mode:logical Min. : 4.00
## NA's:1181 1st Qu.:0 Linda :176 NA's:1181 1st Qu.: 5.00
## Median :0 mailin :120 Median :18.00
## Mean :0 Mailin : 88 Mean :16.23
## 3rd Qu.:0 Susanne:152 3rd Qu.:24.00
## Max. :0 Y :420 Max. :65.00
## yuqiu : 45
## months gender
## Min. : 0.000 m:292
## 1st Qu.: 0.000 w:889
## Median : 0.000
## Mean : 2.767
## 3rd Qu.: 6.000
## Max. :11.000
## Was ist zu tun?
- leere und überflüssige Spalten entfernen
- welche sind das?
- Faktoren codieren
- V8 enthält alle Informationen für die Faktoren und
- die Spalte age enthält die Informationen für
- Spalten umbenennen
Leere und überflüssige Spalten entfernen
Spalten auswählen, die wir behalten wollen:
## V1 V2 V3 V4 V5 V6 V7 V8 V9 V10 V11
## 0 0 0 0 0 0 0 0 0 100 0
## V12 V13 V14 V15 V16 V17 age months gender
## 0 100 100 0 0 100 0 0 0## V2 V8 V9 V16 age months gender
## 1 3536fed0235c7b34a33ddf06fb91b390 schaffen4_at 5 mailin 5 1 w
## 2 3536fed0235c7b34a33ddf06fb91b390 schaffen1_non 5 mailin 5 1 w
## 3 3536fed0235c7b34a33ddf06fb91b390 cleft2_non 5 mailin 5 1 w
## 4 3536fed0235c7b34a33ddf06fb91b390 appRel3_non 5 mailin 5 1 w
## 5 3536fed0235c7b34a33ddf06fb91b390 appRel9_non 4 mailin 5 1 w
## 6 3536fed0235c7b34a33ddf06fb91b390 gewinnen2_non 4 mailin 5 1 wLeere und überflüssige Spalten entfernen
Alternative mit subset-Fuktion:
Faktor
Was wir machen müssen, ist nach Mustern in der Itembenennung zu schauen, die wir verwenden können. Dazu bieten sich “_at” und “_non” an. Eine Funktion, die wir uns zunutze machen können, ist im Paket “stringr” enthalten.
Sie heißt str_detect und funktioniert so:
Ein Beispiel:
## [1] FALSE TRUE FALSE FALSEÜbung
- Benutzt
str_detectum eine neue Spalte “issueness” anzulegen, die “at-issue” enthält, wenn das Item den String “_at” enthält, und “non-at-issue” bei “_non”
Lösung
- Benutzt
str_detectum eine neue Spalte “issueness” anzulegen, die “at-issue” enthält, wenn das Item den String “_at” enthält, und “non-at-issue” bei “_non”
d$issueness[str_detect(d$V8, "_at")] <- "at-issue"
d$issueness[str_detect(d$V8, "_non")] <- "non-at-issue"
head(d)## V2 V8 V9 V16 age months gender
## 1 3536fed0235c7b34a33ddf06fb91b390 schaffen4_at 5 mailin 5 1 w
## 2 3536fed0235c7b34a33ddf06fb91b390 schaffen1_non 5 mailin 5 1 w
## 3 3536fed0235c7b34a33ddf06fb91b390 cleft2_non 5 mailin 5 1 w
## 4 3536fed0235c7b34a33ddf06fb91b390 appRel3_non 5 mailin 5 1 w
## 5 3536fed0235c7b34a33ddf06fb91b390 appRel9_non 4 mailin 5 1 w
## 6 3536fed0235c7b34a33ddf06fb91b390 gewinnen2_non 4 mailin 5 1 w
## issueness
## 1 at-issue
## 2 non-at-issue
## 3 non-at-issue
## 4 non-at-issue
## 5 non-at-issue
## 6 non-at-issueFaktor
Die Liste von Triggern sieht wie folgt aus:
- Soft: schaffen, entdecken, gewinnen
- Hard: cleft, auch, wieder
- Appositiver Relativsatz: appRel
Übung II
- Benutzt dieselbe Methode, um den Faktor zu codieren.
Tipp: str_detect erlaubt auch Listen von Mustern, deren Elemente durch “|” (read: oder) innerhalb eines Strings getrennt sind:
fruit <- c("apple", "banana", "pear", "pineapple")
str_detect(fruit, "na|ar") # TRUE bei "na" und "ar"## [1] FALSE TRUE TRUE FALSE## [1] "banana" "pear"Lösung II
- Benutzt dieselbe Methode, um den Faktor zu codieren
d$trigger[str_detect(d$V8, "schaffen|entdecken|gewinnen")] <- "soft"
d$trigger[str_detect(d$V8, "cleft|auch|wieder")] <- "hard"
d$trigger[str_detect(d$V8, "appRel")] <- "appRel"
head(d)## V2 V8 V9 V16 age months gender
## 1 3536fed0235c7b34a33ddf06fb91b390 schaffen4_at 5 mailin 5 1 w
## 2 3536fed0235c7b34a33ddf06fb91b390 schaffen1_non 5 mailin 5 1 w
## 3 3536fed0235c7b34a33ddf06fb91b390 cleft2_non 5 mailin 5 1 w
## 4 3536fed0235c7b34a33ddf06fb91b390 appRel3_non 5 mailin 5 1 w
## 5 3536fed0235c7b34a33ddf06fb91b390 appRel9_non 4 mailin 5 1 w
## 6 3536fed0235c7b34a33ddf06fb91b390 gewinnen2_non 4 mailin 5 1 w
## issueness trigger
## 1 at-issue soft
## 2 non-at-issue soft
## 3 non-at-issue hard
## 4 non-at-issue appRel
## 5 non-at-issue appRel
## 6 non-at-issue softFaktor
Jetzt müssen wir noch die letzte unabhängige Variable kodieren und der schwerste Teil ist geschafft
Übung:
- Erstellt den Faktor “stage” im Datenblatt mit den Stufen “adult” und “child”
Lösung III
## V2 V8 V9 V16 age months gender
## 1 3536fed0235c7b34a33ddf06fb91b390 schaffen4_at 5 mailin 5 1 w
## 2 3536fed0235c7b34a33ddf06fb91b390 schaffen1_non 5 mailin 5 1 w
## 3 3536fed0235c7b34a33ddf06fb91b390 cleft2_non 5 mailin 5 1 w
## 4 3536fed0235c7b34a33ddf06fb91b390 appRel3_non 5 mailin 5 1 w
## 5 3536fed0235c7b34a33ddf06fb91b390 appRel9_non 4 mailin 5 1 w
## 6 3536fed0235c7b34a33ddf06fb91b390 gewinnen2_non 4 mailin 5 1 w
## issueness trigger stage
## 1 at-issue soft child
## 2 non-at-issue soft child
## 3 non-at-issue hard child
## 4 non-at-issue appRel child
## 5 non-at-issue appRel child
## 6 non-at-issue soft childLetzte Schritte
Wir haben es fast geschafft! Es bleiben noch zwei Dinge zu tun.
Übung:
- Benennt die Spalten sinnvoll um
- Überprüft, ob die wichtigen Spalten Faktoren sind und holt das ggf nach
Lösung IV
- Benennt die Spalten sinnvoll um
Lösung IV
- Überprüft, ob die wichtigen Spalten Faktoren sind und holt das ggf nach
## 'data.frame': 1181 obs. of 10 variables:
## $ id : Factor w/ 41 levels "071362c17b307af0925ac2d94d697527",..: 6 6 6 6 6 6 6 6 6 6 ...
## $ item : Factor w/ 59 levels "appRel1_at","appRel1_non",..: 52 47 29 8 20 43 57 23 33 2 ...
## $ judgment : int 5 5 5 5 4 4 3 4 5 4 ...
## $ tester : Factor w/ 7 levels "Imke","Linda",..: 3 3 3 3 3 3 3 3 3 3 ...
## $ years : int 5 5 5 5 5 5 5 5 5 5 ...
## $ months : int 1 1 1 1 1 1 1 1 1 1 ...
## $ gender : Factor w/ 2 levels "m","w": 2 2 2 2 2 2 2 2 2 2 ...
## $ issueness: chr "at-issue" "non-at-issue" "non-at-issue" "non-at-issue" ...
## $ trigger : chr "soft" "soft" "hard" "appRel" ...
## $ stage : chr "child" "child" "child" "child" ...d$issueness <- factor(d$issueness)
d$trigger <- factor(d$trigger)
d$stage <- factor(d$stage)
str(d[, 8:10])## 'data.frame': 1181 obs. of 3 variables:
## $ issueness: Factor w/ 2 levels "at-issue","non-at-issue": 1 2 2 2 2 2 2 1 2 2 ...
## $ trigger : Factor w/ 3 levels "appRel","hard",..: 3 3 2 1 1 3 2 2 2 1 ...
## $ stage : Factor w/ 2 levels "adult","child": 2 2 2 2 2 2 2 2 2 2 ...(Optionale Kür)
Um die spätere Analyse ein bisschen einfacher (und die by-item-Plots aufschlussreicher) zu machen, codieren wir im Folgenden noch die Spalte mit den Itemids (also ohne die At-Issueness-Information) und die lexikalischen Trigger (Wert in der Itemspalte ohne Zahl und ohne At-Issueness)
# generate a column that holds the item ids without at-issuess
# replace strings like "_at" with nothing
d$itemid <- factor(gsub("_.*$", "", d$item))
# code lexical triggers
# replace strings like "5_non" with nothing
d$lex <- factor(gsub("[[:digit:]]_.*$", "", d$item))
head(d, 4)## id item judgment tester years months
## 1 3536fed0235c7b34a33ddf06fb91b390 schaffen4_at 5 mailin 5 1
## 2 3536fed0235c7b34a33ddf06fb91b390 schaffen1_non 5 mailin 5 1
## 3 3536fed0235c7b34a33ddf06fb91b390 cleft2_non 5 mailin 5 1
## 4 3536fed0235c7b34a33ddf06fb91b390 appRel3_non 5 mailin 5 1
## gender issueness trigger stage itemid lex
## 1 w at-issue soft child schaffen4 schaffen
## 2 w non-at-issue soft child schaffen1 schaffen
## 3 w non-at-issue hard child cleft2 cleft
## 4 w non-at-issue appRel child appRel3 appRelDeskriptive Statistik
describe()/describeBy()
Für die descriptive Statistik können wir der Einfachheit halber den describeBy-Befehl verwenden
Paket laden:
Statistik rechnen lassen:
Lösungen V
## item group1 group2 group3 vars n mean sd median trimmed mad min
## X11 1 adult at-issue appRel 1 99 1.85 0.83 2 1.77 1.48 1
## X12 2 child at-issue appRel 1 99 3.36 1.45 4 3.44 1.48 1
## X13 3 adult non-at-issue appRel 1 100 3.99 0.95 4 4.12 1.48 1
## X14 4 child non-at-issue appRel 1 100 3.71 1.32 4 3.86 1.48 1
## X15 5 adult at-issue hard 1 100 1.99 1.01 2 1.85 1.48 1
## X16 6 child at-issue hard 1 94 3.39 1.48 4 3.49 1.48 1
## X17 7 adult non-at-issue hard 1 97 4.33 1.01 5 4.53 0.00 1
## X18 8 child non-at-issue hard 1 98 3.80 1.32 4 3.96 1.48 1
## X19 9 adult at-issue soft 1 100 3.48 1.18 4 3.51 1.48 1
## X110 10 child at-issue soft 1 97 3.70 1.35 4 3.85 1.48 1
## X111 11 adult non-at-issue soft 1 100 4.18 1.01 4 4.38 1.48 1
## X112 12 child non-at-issue soft 1 97 3.84 1.34 4 4.01 1.48 1
## max range skew kurtosis se
## X11 4 3 0.71 -0.13 0.08
## X12 5 4 -0.28 -1.34 0.15
## X13 5 4 -0.97 0.44 0.09
## X14 5 4 -0.64 -0.84 0.13
## X15 5 4 0.95 0.08 0.10
## X16 5 4 -0.27 -1.44 0.15
## X17 5 4 -1.65 2.24 0.10
## X18 5 4 -0.79 -0.66 0.13
## X19 5 4 -0.28 -1.15 0.12
## X110 5 4 -0.62 -0.94 0.14
## X111 5 4 -1.53 2.15 0.10
## X112 5 4 -0.82 -0.66 0.14Plots
Plan
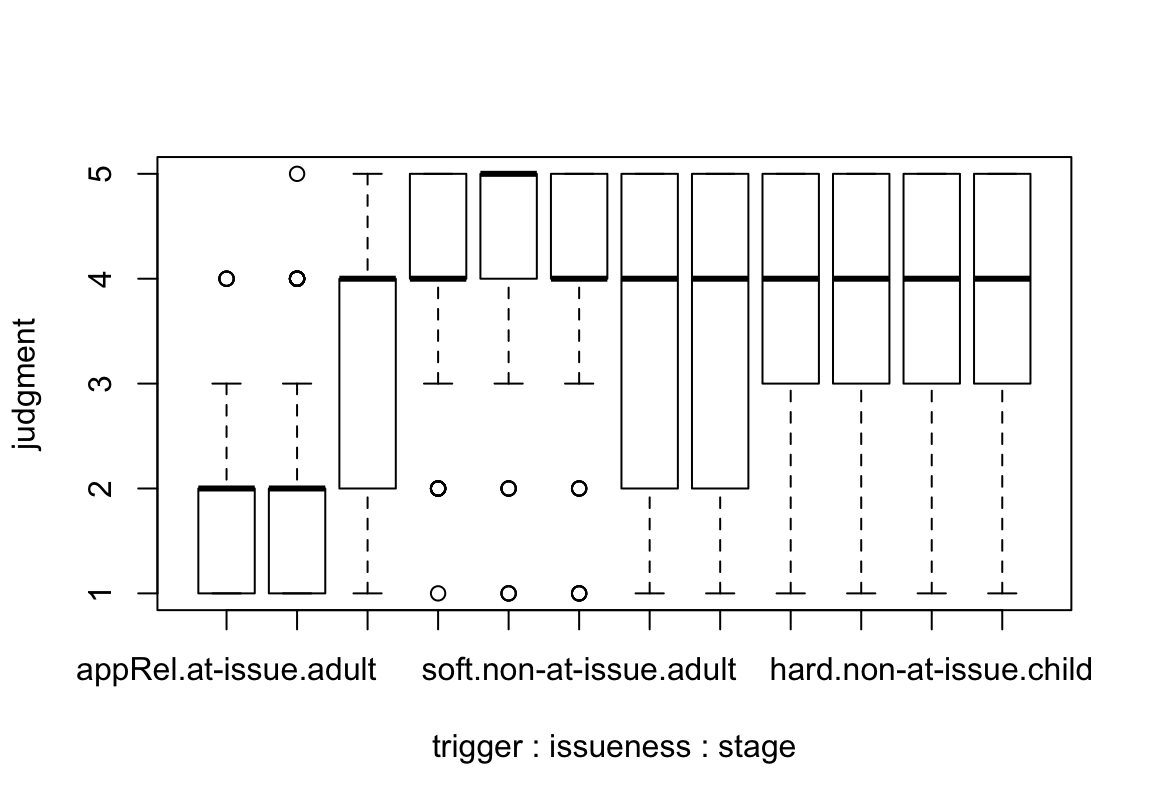
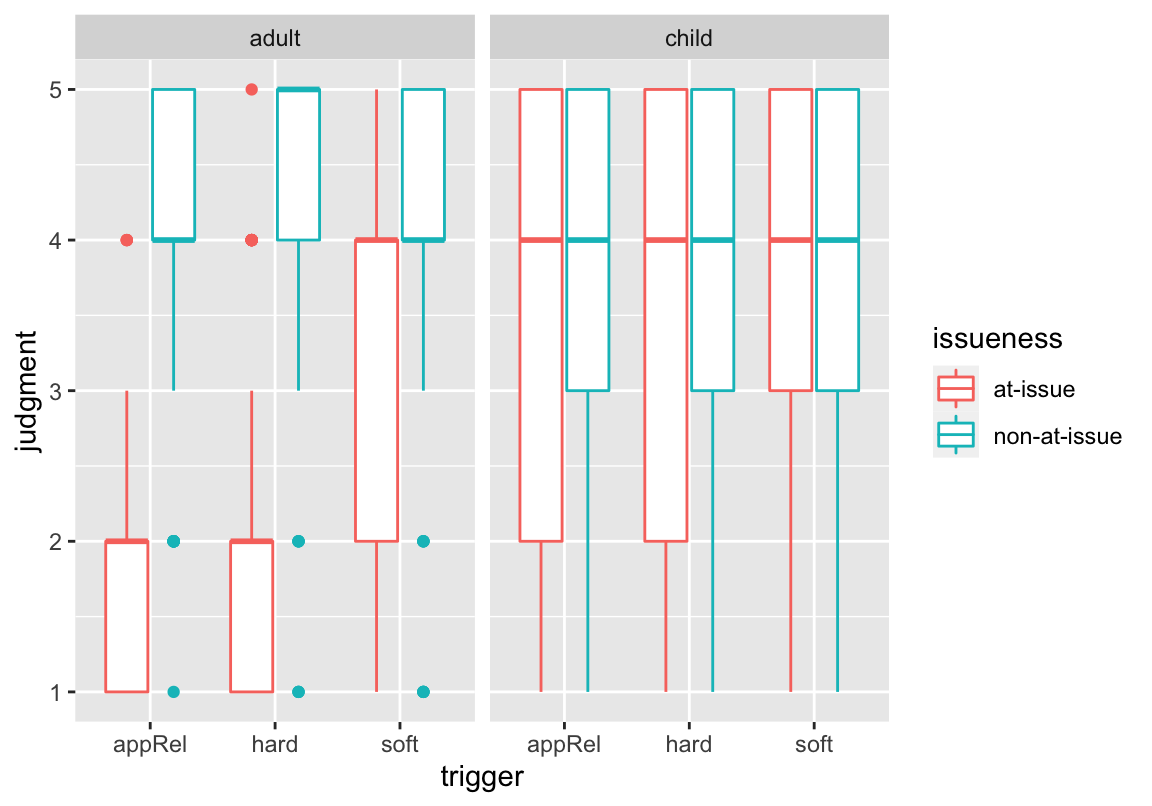
Im Folgenden zeige ich euch zwei Boxplots der Daten, die dieselben Informationen zeigen. Nämlich wie sich die unterschiedlichen Faktoren (bzw deren Stufen) auf die Bewertungen auswirken
Zuerst kommt der boxplot-Befehl von R. Der Plot wird wenig übersichtlich sein.
Danach zeige ich euch einen Plot mit ggplot2, der wesentlich besser verständlich ist. Dazu brauchen wir Folgendens:
Inferenzstatistik
Für einen Test entscheiden
Welchen Test müssen wir rechnen?
Wir müssen folgende Faktoren in unserer Analyse unterbringen und schauen, ob und wie sie sich auf die abhängige Variable (“judgment” – Akzeptabilitätsurteile) auswirkgen:
Welcher Test bietet sich an?
Lösung
Die Varianzanalyse!
nächste Übung: schreibt einen Befehl für die Varianzanlyse mit dem afex-Paket. Falls ihr Hilfe braucht, schaut gerne in die vorherigen Folien oder gebt ?aov_ez ein, nachdem ihr das Paket geladen habt.
Lösung
Lösung
## Anova Table (Type 3 tests)
##
## Response: judgment
## Effect df MSE F ges p.value
## 1 stage 1, 39 1.77 3.56 + .043 .067
## 2 issueness 1, 39 0.33 190.63 *** .312 <.001
## 3 stage:issueness 1, 39 0.33 96.53 *** .187 <.001
## 4 trigger 1.88, 73.25 0.42 17.35 *** .089 <.001
## 5 stage:trigger 1.88, 73.25 0.42 7.36 ** .040 .002
## 6 issueness:trigger 1.85, 72.03 0.37 15.51 *** .071 <.001
## 7 stage:issueness:trigger 1.85, 72.03 0.37 9.22 *** .043 <.001
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1
##
## Sphericity correction method: GGLösung
## Anova Table (Type 3 tests)
##
## Response: judgment
## Effect df MSE F ges p.value
## 1 trigger 2, 26 0.22 15.80 *** .298 <.001
## 2 issueness 1, 26 0.21 145.05 *** .644 <.001
## 3 trigger:issueness 2, 26 0.21 12.64 *** .240 <.001
## 4 stage 1, 26 0.05 89.51 *** .214 <.001
## 5 trigger:stage 2, 26 0.05 21.52 *** .116 <.001
## 6 issueness:stage 1, 26 0.16 84.99 *** .446 <.001
## 7 trigger:issueness:stage 2, 26 0.16 7.65 ** .127 .002
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1Für ausführlichere Wiedergabe der ANOVA
##
## Univariate Type III Repeated-Measures ANOVA Assuming Sphericity
##
## Sum Sq num Df Error SS den Df F value
## (Intercept) 2950.73 1 69.078 39 1665.9223
## stage 6.31 1 69.078 39 3.5631
## issueness 63.03 1 12.896 39 190.6296
## stage:issueness 31.92 1 12.896 39 96.5296
## trigger 13.53 2 30.407 78 17.3550
## stage:trigger 5.74 2 30.407 78 7.3608
## issueness:trigger 10.60 2 26.661 78 15.5099
## stage:issueness:trigger 6.30 2 26.661 78 9.2192
## Pr(>F)
## (Intercept) < 0.00000000000000022 ***
## stage 0.0665353 .
## issueness < 0.00000000000000022 ***
## stage:issueness 0.000000000004209 ***
## trigger 0.000000582315820 ***
## stage:trigger 0.0011793 **
## issueness:trigger 0.000002132872576 ***
## stage:issueness:trigger 0.0002546 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
##
## Mauchly Tests for Sphericity
##
## Test statistic p-value
## trigger 0.93521 0.28007
## stage:trigger 0.93521 0.28007
## issueness:trigger 0.91718 0.19347
## stage:issueness:trigger 0.91718 0.19347
##
##
## Greenhouse-Geisser and Huynh-Feldt Corrections
## for Departure from Sphericity
##
## GG eps Pr(>F[GG])
## trigger 0.93915 0.000001154 ***
## stage:trigger 0.93915 0.0015242 **
## issueness:trigger 0.92351 0.000004587 ***
## stage:issueness:trigger 0.92351 0.0003904 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## HF eps Pr(>F[HF])
## trigger 0.9850323 0.0000006888931
## stage:trigger 0.9850323 0.0012560891080
## issueness:trigger 0.9673658 0.0000029565151
## stage:issueness:trigger 0.9673658 0.0003055377807Exkurs: Sphärizitätskorrekturen
“In particular, RM ANOVA assumes sphericity. That is, the variances of the differences between all pairs of groups are equal. In repeated measures data, this would imply that the variance of e.g. time 1 and time 2 is the same as the variance of the difference of time 1 and time 3.” Peter Flom (2014)
Sphärizität in der afex-ANOVA
Die afex-ANOVA korrigiert automatisch die Freiheitsgrade (und damit den p-Wert) bei Sphärizitätsverletzungen. Dies kann wie folgt ausgestellt werden:
Vergleich beider Ergebnisse
## Anova Table (Type 3 tests)
##
## Response: judgment
## Effect df MSE F ges p.value
## 1 stage 1, 39 1.77 3.56 + .043 .067
## 2 issueness 1, 39 0.33 190.63 *** .312 <.001
## 3 stage:issueness 1, 39 0.33 96.53 *** .187 <.001
## 4 trigger 1.88, 73.25 0.42 17.35 *** .089 <.001
## 5 stage:trigger 1.88, 73.25 0.42 7.36 ** .040 .002
## 6 issueness:trigger 1.85, 72.03 0.37 15.51 *** .071 <.001
## 7 stage:issueness:trigger 1.85, 72.03 0.37 9.22 *** .043 <.001
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1
##
## Sphericity correction method: GG## Anova Table (Type 3 tests)
##
## Response: judgment
## Effect df MSE F ges p.value
## 1 stage 1, 39 1.77 3.56 + .043 .067
## 2 issueness 1, 39 0.33 190.63 *** .312 <.001
## 3 stage:issueness 1, 39 0.33 96.53 *** .187 <.001
## 4 trigger 2, 78 0.39 17.35 *** .089 <.001
## 5 stage:trigger 2, 78 0.39 7.36 ** .040 .001
## 6 issueness:trigger 2, 78 0.34 15.51 *** .071 <.001
## 7 stage:issueness:trigger 2, 78 0.34 9.22 *** .043 <.001
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '+' 0.1 ' ' 1Kontraste
Kontraste kodieren, aber wie?
library(emmeans)
# referenztabelle für kontraste; specs sind die die Faktoren, dessen estimated marginal means (EMM) wir uns angucken wollen
(ref_id <- emmeans(anovasub,
specs = c("stage", "trigger", "issueness")))## stage trigger issueness emmean SE df lower.CL upper.CL
## adult appRel at.issue 1.85 0.171 133 1.51 2.19
## child appRel at.issue 3.34 0.169 129 3.01 3.68
## adult hard at.issue 1.99 0.171 133 1.66 2.33
## child hard at.issue 3.46 0.169 129 3.12 3.79
## adult soft at.issue 3.48 0.171 133 3.15 3.82
## child soft at.issue 3.65 0.169 129 3.31 3.98
## adult appRel non.at.issue 3.99 0.171 133 3.66 4.33
## child appRel non.at.issue 3.67 0.169 129 3.34 4.01
## adult hard non.at.issue 4.35 0.171 133 4.01 4.69
## child hard non.at.issue 3.84 0.169 129 3.51 4.18
## adult soft non.at.issue 4.18 0.171 133 3.85 4.52
## child soft non.at.issue 3.81 0.169 129 3.47 4.14
##
## Warning: EMMs are biased unless design is perfectly balanced
## Confidence level used: 0.95Exkurs: Estimated Marginal Means
# let's generate a table with values
d_test <- tibble(
reads = c("reads", "doesnt_read"),
studies = c(1, 2),
doesnt_study = c(3, 5)
)
d_test## # A tibble: 2 x 3
## reads studies doesnt_study
## <chr> <dbl> <dbl>
## 1 reads 1 3
## 2 doesnt_read 2 5# then lets transform the data into a tidy one (one row = one observation)
d_test <- d_test %>%
pivot_longer(c("studies", "doesnt_study"),
names_to = "prepares",
values_to = "grade")
d_test## # A tibble: 4 x 3
## reads prepares grade
## <chr> <chr> <dbl>
## 1 reads studies 1
## 2 reads doesnt_study 3
## 3 doesnt_read studies 2
## 4 doesnt_read doesnt_study 5Exkurs cont’d
# then lets fit a model
fit <- aov(grade ~ reads * prepares, data = d_test)
# and compute estimated marginal means
# in a balanced design, EMMs just represent the cell means (here, because we only have one observation per factor combination, means = observations)
# in unbalanced design EMMs are adjusted so that all cell means are given equal weight
(ref <- emmeans(fit, c("reads", "prepares")))## Warning in qt((1 - level)/adiv, df): NaNs produced## reads prepares emmean SE df lower.CL upper.CL
## doesnt_read doesnt_study 5 NaN 0 NaN NaN
## reads doesnt_study 3 NaN 0 NaN NaN
## doesnt_read studies 2 NaN 0 NaN NaN
## reads studies 1 NaN 0 NaN NaN
##
## Confidence level used: 0.95Übung
Spezifiziert Vektoren für die folgenden Kontraste mithilfe der Referenztabelle:
- appositive RCs (at-issue) vs Hard Trigger (at-issue)
- Soft Trigger (at-issue) vs Hard Trigger (at-issue)
- At-issue, Kinder vs At-issue, Erwachsene
Beispiel: um adult_appRel mit child_appRel (und nichts anderes) zu vergleichen, verwenden wir folgende Spezifizierung:
Lösung
Spezifiziert Vektoren für die folgenden Kontraste mithilfe der Referenztabelle:
- appositive RCs (at-issue) vs Hard Trigger (at-issue)
- Soft Trigger (at-issue) vs Hard Trigger (at-issue)
- At-issue, Kinder vs At-issue, Erwachsene
Kontraste berechnen
Bonferronikorrektur nur für post hoc Kontraste absolut nötig. Bei a priori spezifizierten (das heißt von der Hypothese gedeckten) Kontrasten kann diese Korrektur weggelassen werden.
# bonferroni-korrigierte tests berechnen
summary(contrast(ref_id, list(RCvHTat = test_RCvHTat,
STvHTat = test_STvHTat,
ATcvATa = test_ATcvATa),
adjust = "bonferroni", # Korrektur für multiple Tests
infer = TRUE # zeigt p-Werte und KonfInt
))## contrast estimate SE df lower.CL upper.CL t.ratio p.value
## RCvHTat -0.259 0.267 155.3 -0.906 0.387 -0.971 0.9997
## STvHTat 1.680 0.267 155.3 1.034 2.327 6.288 <.0001
## ATcvATa 3.123 0.555 53.1 1.752 4.495 5.630 <.0001
##
## Confidence level used: 0.95
## Conf-level adjustment: bonferroni method for 3 estimates
## P value adjustment: bonferroni method for 3 testsZum selbst Ausprobieren
Warning von Thomas (pc): “D.h. ich wuerde den Leuten sagen, dass der letzte Code-Schnipsel (wo Du dann alle Vergleiche anschaust) eine Mischung aus prae- und post-hoc-Betrachtung ist. Und dass das natuerlich fuer moralisch sproede bzw. vom publication bias und Befristung weichgeklopfte Gemueter gewisse Verlockungen/Gefahren birgt. You see?”
# alle paarweisen vergleiche für die beiden level von issueness berechnen
ref_big <- emmeans(anovasub, ~ stage * trigger | issueness)
ref_big## issueness = at.issue:
## stage trigger emmean SE df lower.CL upper.CL
## adult appRel 1.85 0.171 133 1.51 2.19
## child appRel 3.34 0.169 129 3.01 3.68
## adult hard 1.99 0.171 133 1.66 2.33
## child hard 3.46 0.169 129 3.12 3.79
## adult soft 3.48 0.171 133 3.15 3.82
## child soft 3.65 0.169 129 3.31 3.98
##
## issueness = non.at.issue:
## stage trigger emmean SE df lower.CL upper.CL
## adult appRel 3.99 0.171 133 3.66 4.33
## child appRel 3.67 0.169 129 3.34 4.01
## adult hard 4.35 0.171 133 4.01 4.69
## child hard 3.84 0.169 129 3.51 4.18
## adult soft 4.18 0.171 133 3.85 4.52
## child soft 3.81 0.169 129 3.47 4.14
##
## Warning: EMMs are biased unless design is perfectly balanced
## Confidence level used: 0.95Ergebnisse des großen Paarvergleichs
## issueness = at.issue:
## contrast estimate SE df lower.CL upper.CL t.ratio
## adult,appRel - child,appRel -1.4980 0.241 131 -2.218 -0.7779 -6.220
## adult,appRel - adult,hard -0.1475 0.191 155 -0.718 0.4227 -0.771
## adult,appRel - child,hard -1.6099 0.241 131 -2.330 -0.8898 -6.684
## adult,appRel - adult,soft -1.6375 0.191 155 -2.208 -1.0673 -8.561
## adult,appRel - child,soft -1.8004 0.241 131 -2.520 -1.0802 -7.475
## child,appRel - adult,hard 1.3505 0.241 131 0.630 2.0706 5.607
## child,appRel - child,hard -0.1119 0.187 155 -0.668 0.4446 -0.600
## child,appRel - adult,soft -0.1395 0.241 131 -0.860 0.5806 -0.579
## child,appRel - child,soft -0.3024 0.187 155 -0.859 0.2541 -1.620
## adult,hard - child,hard -1.4624 0.241 131 -2.182 -0.7423 -6.072
## adult,hard - adult,soft -1.4900 0.191 155 -2.060 -0.9198 -7.790
## adult,hard - child,soft -1.6529 0.241 131 -2.373 -0.9327 -6.863
## child,hard - adult,soft -0.0276 0.241 131 -0.748 0.6925 -0.115
## child,hard - child,soft -0.1905 0.187 155 -0.747 0.3660 -1.020
## adult,soft - child,soft -0.1629 0.241 131 -0.883 0.5573 -0.676
## p.value
## <.0001
## 1.0000
## <.0001
## <.0001
## <.0001
## <.0001
## 1.0000
## 1.0000
## 1.0000
## <.0001
## <.0001
## <.0001
## 1.0000
## 1.0000
## 1.0000
##
## issueness = non.at.issue:
## contrast estimate SE df lower.CL upper.CL t.ratio
## adult appRel - child appRel 0.3194 0.241 131 -0.401 1.0395 1.326
## adult appRel - adult hard -0.3525 0.191 155 -0.923 0.2177 -1.843
## adult appRel - child hard 0.1519 0.241 131 -0.568 0.8720 0.631
## adult appRel - adult soft -0.1900 0.191 155 -0.760 0.3802 -0.993
## adult appRel - child soft 0.1868 0.241 131 -0.533 0.9069 0.776
## child appRel - adult hard -0.6719 0.241 131 -1.392 0.0482 -2.790
## child appRel - child hard -0.1675 0.187 155 -0.724 0.3890 -0.897
## child appRel - adult soft -0.5094 0.241 131 -1.229 0.2107 -2.115
## child appRel - child soft -0.1325 0.187 155 -0.689 0.4239 -0.710
## adult hard - child hard 0.5044 0.241 131 -0.216 1.2245 2.094
## adult hard - adult soft 0.1625 0.191 155 -0.408 0.7327 0.850
## adult hard - child soft 0.5393 0.241 131 -0.181 1.2594 2.239
## child hard - adult soft -0.3419 0.241 131 -1.062 0.3782 -1.420
## child hard - child soft 0.0349 0.187 155 -0.522 0.5914 0.187
## adult soft - child soft 0.3768 0.241 131 -0.343 1.0969 1.565
## p.value
## 1.0000
## 1.0000
## 1.0000
## 1.0000
## 1.0000
## 0.0910
## 1.0000
## 0.5449
## 1.0000
## 0.5723
## 1.0000
## 0.4023
## 1.0000
## 1.0000
## 1.0000
##
## Confidence level used: 0.95
## Conf-level adjustment: bonferroni method for 15 estimates
## P value adjustment: bonferroni method for 15 testsNochmal Plots
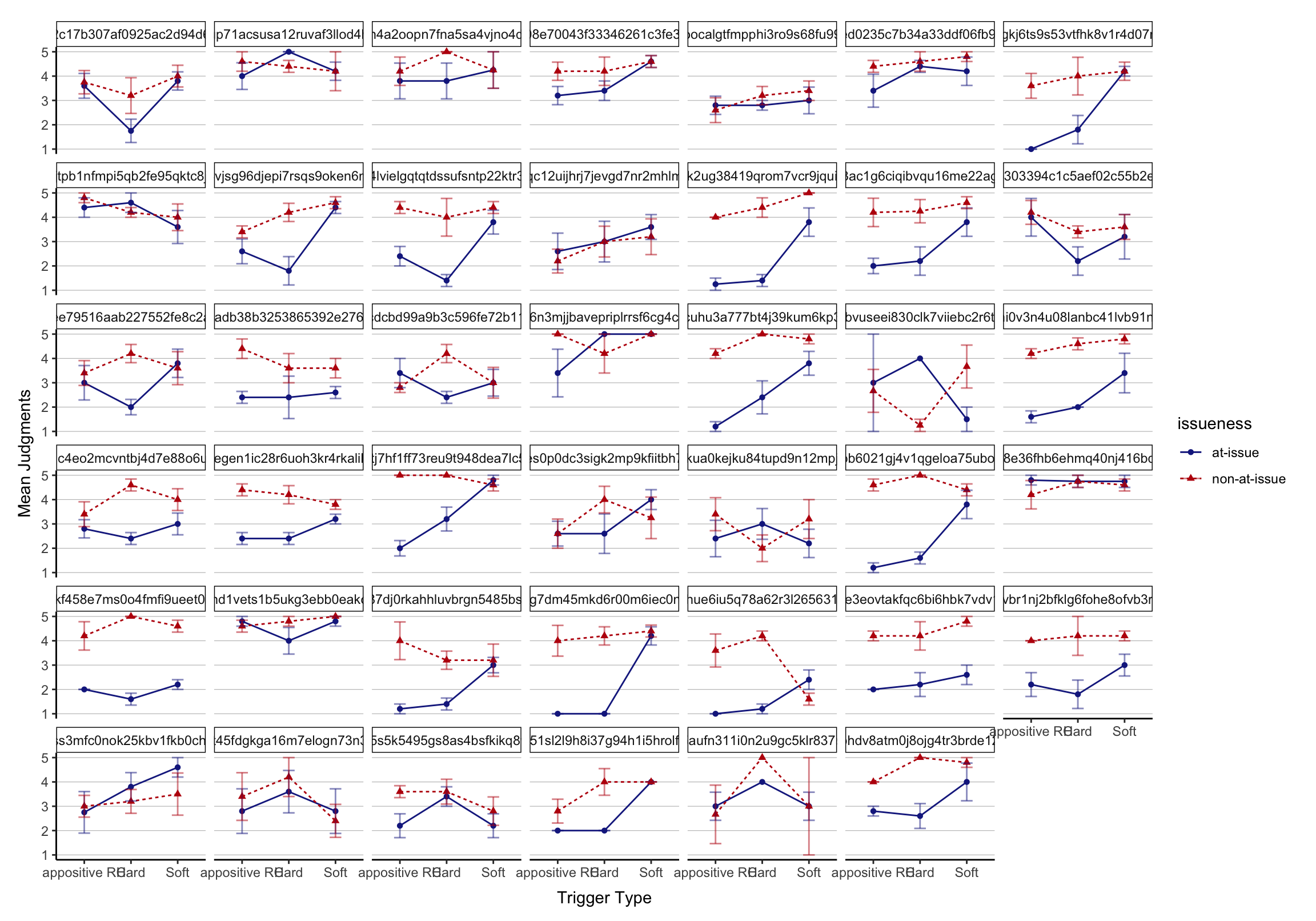
by subject
subs <- ggplot(d, aes(y = judgment, x = trigger, color = issueness,
shape = issueness, linetype = issueness,
group = issueness)) +
stat_summary(fun.y = mean, geom = "line") +
stat_summary(fun.y = mean, geom = "point") +
stat_summary(fun.data = mean_se, geom = "errorbar", width = 0.25,
alpha = .5, linetype = 1) +
labs(y = "Mean Judgments", x = "Trigger Type") +
scale_color_manual(values = c("#19278e", "#BC0D0D")) +
scale_x_discrete(labels = c('appositive RC', 'Hard', 'Soft')) +
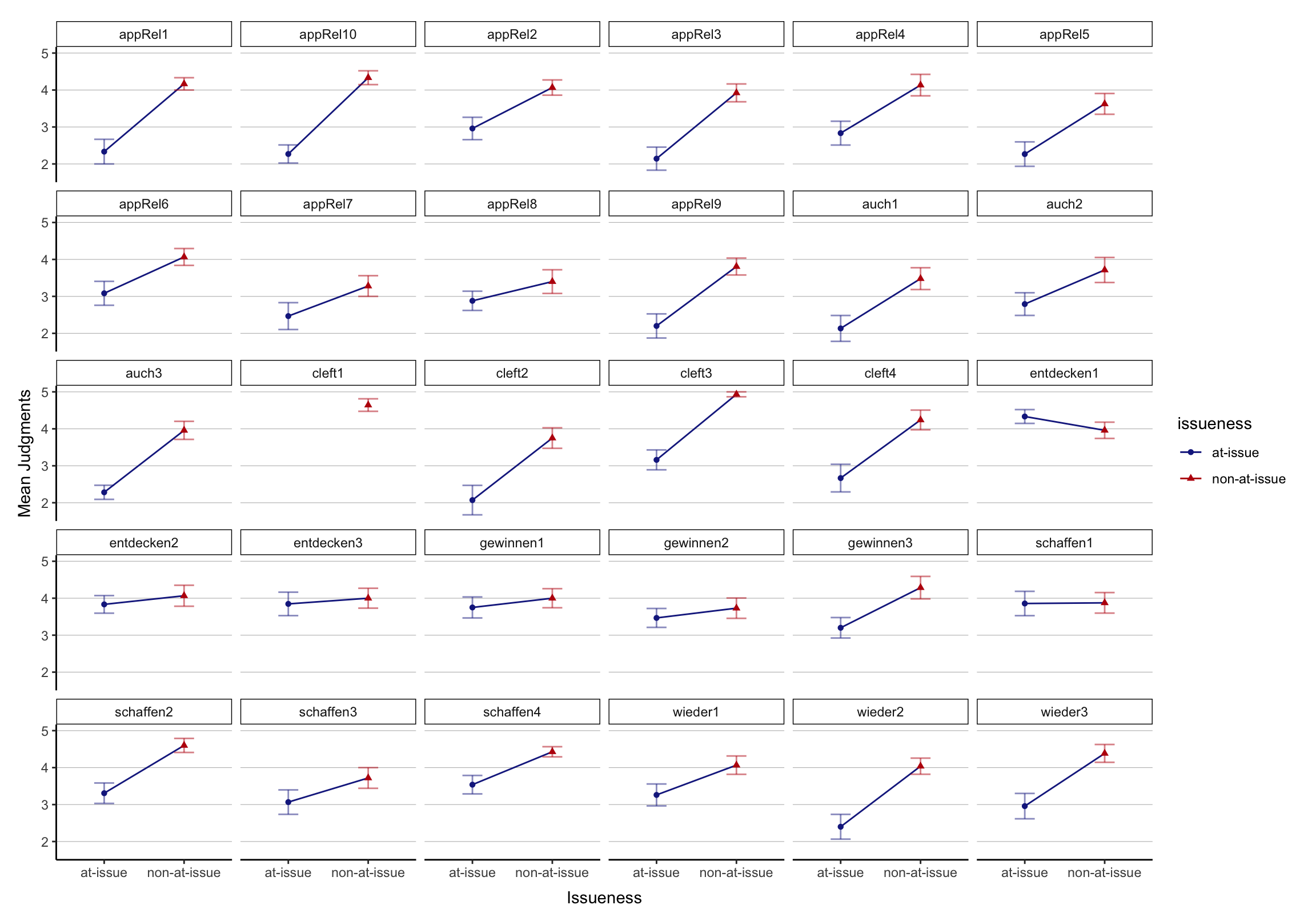
facet_wrap(~id) + cleanupby item
items <- ggplot(d, aes(y = judgment, x = issueness, color = issueness,
shape = issueness)) +
stat_summary(fun.y = mean, geom = "line", aes(group = 1)) +
stat_summary(fun.y = mean, geom = "point", aes(group = 1)) +
stat_summary(fun.data = mean_se, geom = "errorbar", width = 0.25,
alpha = .5, linetype = 1) +
labs(y = "Mean Judgments", x = "Issueness") +
scale_color_manual(values = c("#19278e", "#BC0D0D")) +
facet_wrap(~itemid) + cleanupOff to the rave
Bis zum nächsten Mal!